Da grezzo a raffinato un viaggio attraverso la pre-elaborazione dei dati – Parte 1
From raw to refined, a journey through data pre-processing - Part 1
A volte i dati che riceviamo per i nostri compiti di apprendimento automatico non sono in un formato adatto per il codice con Scikit-Learn o altre librerie di apprendimento automatico. Di conseguenza, dobbiamo elaborare i dati per trasformarli nel formato desiderato.
Potrebbero esserci vari problemi con i dati grezzi. In base alla natura del problema, dobbiamo utilizzare metodi appropriati per affrontarlo.
Vediamo alcuni di questi metodi e come implementarli in un codice.
Rimozione della media e ridimensionamento della varianza (Standardizzazione)
Gli estimatori di Scikit-Learn (Gli estimatori si riferiscono alle classi di Scikit-Learn utilizzate per addestrare i modelli di apprendimento automatico) sono tarati per funzionare al meglio con i dati distribuiti in modo normale standard, cioè una distribuzione gaussiana con media zero e varianza unitaria.
I dati grezzi potrebbero non essere sempre distribuiti in modo gaussiano e quindi i modelli addestrati su questi dati potrebbero fornire risultati sub-ottimali. L’operazione di standardizzazione potrebbe essere la soluzione a questo problema.
La standardizzazione viene eseguita utilizzando la formula seguente:

Prima viene calcolata la media e la deviazione standard per una colonna. Quindi, dalla colonna viene sottratta la media. Infine, il risultato delle sottrazioni viene diviso per la deviazione standard.
Vediamo come implementarlo in un codice.
Per la dimostrazione, utilizziamo il famoso dataset ‘Tips’. Il dataset ‘Tips’ viene utilizzato per prevedere le mance ricevute dai camerieri in base a diversi fattori come il totale del conto, il genere del cliente, il giorno della settimana, l’ora del giorno, ecc.
## Importazione delle librerie richiesteimport warningswarnings.filterwarnings('ignore')import numpy as npimport pandas as pdimport matplotlib.pyplot as pltimport seaborn as sns%matplotlib inline
## Caricamento dei dati.df = pd.read_csv('https://raw.githubusercontent.com/mwaskom/seaborn-data/master/tips.csv')## Visualizzazione di alcune righe dei datidf.head()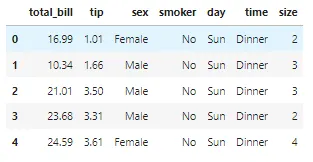
Prima di tutto, dobbiamo separare la caratteristica dipendente, che è ‘total_bill’, dalle caratteristiche indipendenti. Dopo di che, dobbiamo dividere i dati in set di addestramento e di test.
## Importazione del metodo richiesto.from sklearn.model_selection import train_test_split## Separazione delle caratteristiche dipendenti e indipendentiX, y = df.drop('total_bill', axis=1), df['total_bill']## Separazione dei dati di addestramento e di testX_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)Eseguiamo la standardizzazione sulla colonna ‘tip’.
from sklearn.preprocessing import StandardScaler## calcolo della media e della deviazione standard della colonna.scaler = StandardScaler().fit(np.array(X_train['tip']).reshape(-1,1))## trasformazione dei dati utilizzando la media e la deviazione standard calcolatetips_transformed = scaler.transform(np.array(X_test['tip']).reshape(-1,1))tips_transformed[:10]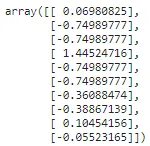
Il metodo ‘reshape’ viene utilizzato per convertire l’array 1D in un array 2D poiché il metodo fit e transform richiede un array 2D come input.
Scaling delle caratteristiche in un intervallo (MinMaxScaler e MaxAbsScaler)
Questa è un’altra approccio alla standardizzazione in cui le caratteristiche vengono ridimensionate in modo che si trovino tra il valore minimo e il valore massimo, spesso tra zero e uno, oppure in modo che il valore assoluto massimo di ogni caratteristica sia ridimensionato ad una dimensione unitaria.
Se vogliamo ridimensionare i nostri dati in modo che si trovino tra i valori ‘min’ e ‘max’ utilizzando MinMaxScaler, viene utilizzata la seguente formula:

MinMaxScaler:
from sklearn.preprocessing import MinMaxScaler## calcolo della media e della deviazione standard della colonna mm_scaler = MinMaxScaler().fit(np.array(X_train['tip']).reshape(-1,1))## trasformazione dei dati utilizzando la media e la deviazione standard calcolate tips_transformed = mm_scaler.transform(np.array(X_test['tip']).reshape(-1,1))tips_transformed[:10]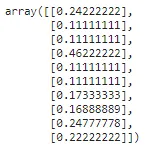
MaxAbsScaler funziona in modo simile, ma scala i dati in modo che ogni valore si trovi nell’intervallo [-1, 1]. Ciò viene fatto dividendo ogni valore per il valore massimo di ciascuna delle caratteristiche.
La centratura dei dati distrugge la sparsità intrinseca dei dati e in generale, non è una cosa appropriata da fare. Ma in caso in cui le caratteristiche sono su scale diverse, ha senso ridimensionare gli input sparsi. MaxAbsScaler è stato appositamente progettato per lo scopo di scalare dati sparsi ed è un modo raccomandato.
MaxAbsScaler:
from sklearn.preprocessing import MaxAbsScaler## calcolo della media e della deviazione standard della colonna ma_scaler = MaxAbsScaler().fit(np.array(X_train['tip']).reshape(-1,1))## trasformazione dei dati utilizzando la media e la deviazione standard calcolate tips_transformed = ma_scaler.transform(np.array(X_test['tip']).reshape(-1,1))tips_transformed[:10]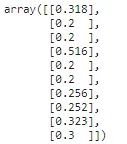
Scaling dei dati con valori anomali (RobustScaler)
Se i dati sono infestati da valori anomali, il valore della media e della deviazione standard potrebbe essere distorto. In questo caso, i valori della media e della deviazione standard non rappresenteranno correttamente il centro dei dati o la loro distribuzione. Pertanto, utilizzare la media e la deviazione standard per la ridimensionamento quando i dati contengono valori anomali non funzionerebbe bene.
Per ovviare a questo problema, possiamo utilizzare RobustScaler, che utilizza stime più robuste per il centro e l’intervallo dei dati.
from sklearn.preprocessing import RobustScaler## calcolo della media e della deviazione standard della colonna r_scaler = RobustScaler().fit(np.array(X_train['tip']).reshape(-1,1))## trasformazione dei dati utilizzando la media e la deviazione standard calcolate tips_transformed = r_scaler.transform(np.array(X_test['tip']).reshape(-1,1))tips_transformed[:10]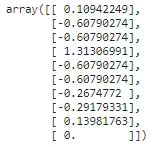
Mappatura su una distribuzione uniforme (QuantileTransformer)
QuantileTransformer può essere utilizzato per mappare i dati su una distribuzione uniforme con valori compresi tra 0 e 1.
from sklearn.preprocessing import QuantileTransformerq_transformer = QuantileTransformer().fit(np.array(X_train['tip']).reshape(-1,1))xtrain_transformed = q_transformer.transform(np.array(X_train['tip']).reshape(-1,1))Ora, visualizziamo la colonna ‘tip’ prima e dopo la trasformazione.
dataframe = pd.DataFrame()dataframe['tips_given'] = X_train['tip']dataframe['tips_uniformDist'] = xtrain_transformedsns.set_style('darkgrid')plt.figure(figsize=(10,5))for index, feature in enumerate(dataframe.columns): plt.subplot(1,2,index+1) sns.distplot(dataframe[feature],kde=True, color='b') plt.xlabel(feature) plt.ylabel('distribuzione') plt.title(f"Distribuzione di {feature}")plt.tight_layout()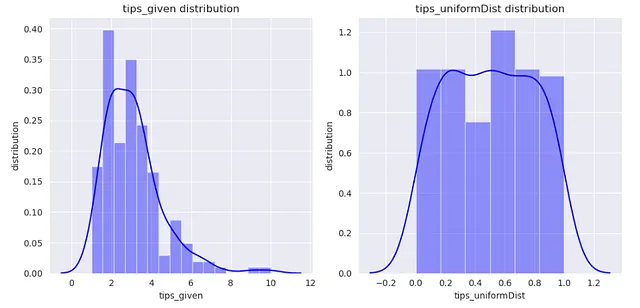
Mappatura su una distribuzione gaussiana (PowerTransformer)
Possiamo utilizzare PowerTransformer per mappare i dati su una distribuzione il più possibile simile a una distribuzione gaussiana.
Possiamo scegliere tra due metodi utilizzati per effettuare questa trasformazione.
- Trasformazione di Box-Cox
- Trasformazione di Yeo-Johnson
Nota che la trasformazione di Box-Cox può essere applicata solo ai dati positivi.
trasformazione di Box-Cox:
from sklearn.preprocessing import PowerTransformer
p_transformer = PowerTransformer(method='box-cox').fit(np.array(X_train['tip']).reshape(-1,1))
xtrain_transformed = p_transformer.transform(np.array(X_train['tip']).reshape(-1,1))
dataframe = pd.DataFrame()
dataframe['tips_given'] = X_train['tip']
dataframe['tips_NormalDist'] = xtrain_transformed
sns.set_style('darkgrid')
plt.figure(figsize=(10,5))
for index, feature in enumerate(dataframe.columns):
plt.subplot(1,2,index+1)
sns.distplot(dataframe[feature],kde=True, color='y')
plt.xlabel(feature)
plt.ylabel('distribuzione')
plt.title(f"Distribuzione di {feature}")
plt.tight_layout()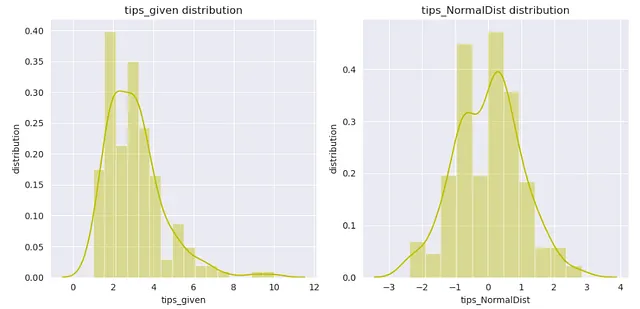
Trasformazione di Yeo-Johnson:
from sklearn.preprocessing import PowerTransformer
p_transformer = PowerTransformer(method='yeo-johnson').fit(np.array(X_train['tip']).reshape(-1,1))
xtrain_transformed = p_transformer.transform(np.array(X_train['tip']).reshape(-1,1))
dataframe = pd.DataFrame()
dataframe['tips_given'] = X_train['tip']
dataframe['tips_NormalDist'] = xtrain_transformed
sns.set_style('darkgrid')
plt.figure(figsize=(10,5))
for index, feature in enumerate(dataframe.columns):
plt.subplot(1,2,index+1)
sns.distplot(dataframe[feature],kde=True, color='g')
plt.xlabel(feature)
plt.ylabel('distribuzione')
plt.title(f"Distribuzione di {feature}")
plt.tight_layout()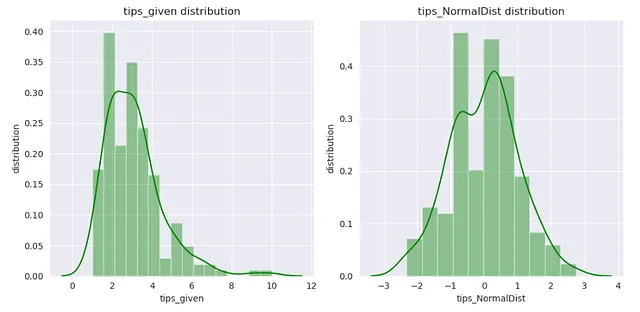
Spero che l’articolo ti piaccia. Se hai qualche pensiero sull’articolo, per favore fammelo sapere. Inoltre, tieni d’occhio il prossimo articolo di questa serie di preelaborazione.
Connettiti con me su
Sito web
Scrivimi all’indirizzo [email protected]
Passa una bella giornata!






